Leukozytenzählung in Kapillarblut-Mikroproben durch gezielte Analyse der DNA-Methylierung mit digitaler Tröpfchen-PCR - "Prick-Me"
In der klinischen Routine ist die Analyse der Leukozyten-Zusammensetzung im peripheren Blut ein wichtiges und täglich angewandtes Verfahren. Die Zellzählung wird in der Regel manuell oder automatisch durchgeführt, aber alle herkömmlichen Methoden erfordern frische Blutproben (nicht älter als 24 Stunden). Alternativ ist es möglich, die zelluläre Zusammensetzung anhand von epigenetischen Modifikationen abzuschätzen, die während der hämatopoetischen Differenzierung erworben werden.
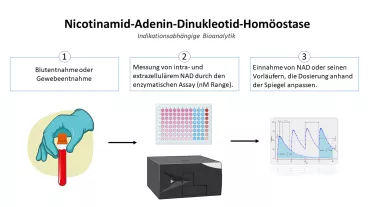
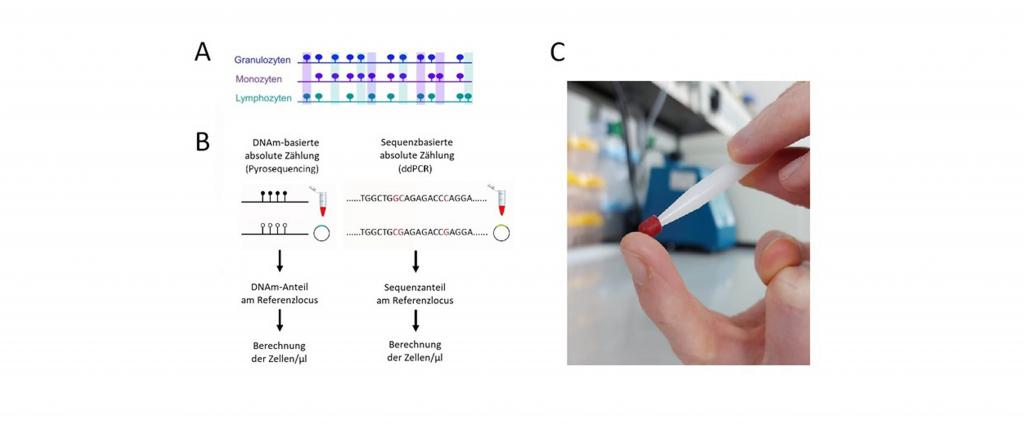
In unseren Vorarbeiten konnten wir zeigen, dass es möglich ist, die DNA-Methylierung (DNAm) an Zelltyp-spezifischen CG-Dinukleotiden (CpGs) zu erfassen, um die absolute und relative Zusammensetzung der weißen Blutkörperchen (WBC) mit Hilfe der sogenanten Pyrosequenzierung abzuschätzen. Epigenetische Zellzählungen sind auch mit gefrorenen Blutproben und kleinen Blutvolumina (ca. 30 µl) möglich.
Das "Prick-Me"-Vorhaben zielt nun darauf ab, diese Anwendung für Kapillarblut, das durch einen einfachen Fingerstich entnommen werden kann, weiterzuentwickeln und zu validieren, und zwar mittels digitaler Tröpfchen-PCR (ddPCR), die als quantitative Methode zuverlässigere Ergebnisse liefert als die Pyrosequenzierung. Das Verfahren wird dadurch weiter vereinfacht und ermöglicht das Sammeln von Proben auch zuhause durch die Patienten selber.
Projektpartner: VIP+ Bundesministerium für Bildung und Forschung (Validierungsförderung VIP+)